J2P4casusTranscriptomics
Transcriptomics voor het identificeren van inflammatoire immuun activiteit bij Reumatoïde Artritis
Berend Veldthuis - berend.veldthuis\@student.nhlstenden.com{.email} - van hall larenstein & NHLstenden
Reumatoïde Artritis (RA) is een chronische auto-immuunziekte die voorkomt bij 5 op de 1000 mensen, voornamelijk bij oudere vrouwen. Het leidt tot langdurige ontstekingen, pijn en stijve gewrichten. Wanneer onbehandeld, kan de ziekte leiden tot permanente schade aan de gewrichten, vorming van noduli en reumatoïde vasculitis, wat kan leiden tot afsterving van bloedvaten[1]. Diagnose gebeurt aan de hand van het ziektebeeld en de aanwezigheid van auto-antistoffen. Wanneer RA vroeg wordt gediagnosticeerd, kunnen klachten met medicatie worden gemanaged[2]. De algemene consensus is dat RA wordt veroorzaakt door een combinatie van genetische aanleg en omgevingsfactoren[6] Veel onderzoeken naar het genetisch aspect trachten specifieke genen en mutaties te koppelen aan RA. Toch is het belangrijk een algemeen beeld te krijgen van het transcriptoom van patiënten met RA[3]. Daaruit volgt de volgende onderzoeksvraag:
Is er een significante verhoging van de genexpressie in het transcriptoom van patiënten met Reumatoïde Artritis vergeleken met gezonde personen?
De volgende deelvragen worden beantwoord:
- Welke genen zijn het sterkst up-gereguleerd bij personen met RA in vergelijking met gezonde personen?
- Tot welke biologische processen behoren deze genen?
- Met welke pathways zijn deze biologische processen geassocieerd?
Methoden
Ter het beantwoorden van de onderzoekvraag zijn er 8 patenten benaderd waarvan 4 gezond en 4 met RA. De aanwezigheid ban RA in de patenten is >12 maanden eerder bepaald met een ACPA-test. Alle samples zijn verkregen met een synoviumbiopt, zie Tabel 1 Voor een overzicht. Het RNA uit de monsters is geïsoleerd en gesequenced.
Tabel1: Overzicht van de 8 monsters. De controlegroep bestaat uit 4 vrouwen met een gemiddelde leeftijd van 30 jaar. De RA-groep bestaat uit 4 vrouwen met een gemiddelde leeftijd van 60 jaar. De gemiddelde leeftijd van alle deelnemers is 45 jaar.
| SampleName | Leeftijd | Groep |
|---|---|---|
| SRR4785819 | 31 | Control |
| SRR4785820 | 15 | Control |
| SRR4785828 | 31 | Control |
| SRR4785831 | 42 | Control |
| SRR4785831 | 42 | Control |
| SRR4785831 | 42 | Control |
| SRR4785979 | 54 | RA |
| SRR4785980 | 66 | RA |
| SRR4785986 | 60 | RA |
| SRR4785988 | 59 | RA |
Om genen met significante veranderingen in expressie (fold change) te identificeren, zijn alle verkregen reads met Rsubread-2.22.1 en Rsamtools-2.24.0 uitgelijnd op het humane referentiegenoom GRCh38.p14.
Deze data is gebruikt om een count matrix te genereren.
Vervolgens is met DESeq2-1.48.1 de genexpressie berekend.
Om biologische processen te koppelen aan de genexpressie is met goseq-1.60.0 en GO.db-3.21.0 een Gene Ontology (GO)-analyse uitgevoerd.
De resultaten hiervan zijn gebruikt om relevante pathways te identificeren op KEGG.jp en te visualiseren met pathview-1.48.0 en KEGGREST-1.48.0.
Zie Figuur 5 oor een flowchart van het proces.
Aanvullend gebruikte packages zijn: BiocManager-1.30.25, readr-2.1.5, tidyverse-2.0.0 en EnhancedVolcano-1.26.0.
---
config:
flowchart:
defaultRenderer: elk
title: Flowchart analyse RA
---
flowchart-elk TB
subgraph thee [Datasets]
HG38[("Genome assembly GRCh38.p14 E.G. <br> *Homo sapiens*(human)")]
dataset[("Dataset of paird end reads <br> .fasta")]
end
subgraph one [Analysis pipeline]
HG38 --> seq_map
HG38 --> matrix
dataset ==> seq_map
seq_map["sequcente mapping <br> • Rsubread-2.22.1; Rsamtools-2.24.0"] ==>|".BAM files"| matrix
matrix["Count Matrix generation <br> • Rsubread-2.22.1; Rsamtools-2.24.0"] ==>|"Count matrix"| data_ana
data_ana["DES analysis <br> • DESeq2-1.48.1; EnhancedVolcano-1.26.0"] ==>|"Fold Change table"| GO_ana
GO_ana["Gene Ontology biologic process analysis <br> • goseq-1.60.0; GO.db-3.21.0"] ==>|"GO:BP processes"| KEGG_ana
KEGG_ana["Pathway analisys using KEGG <br> • pathview-1.48.0; KEGGREST-1.48.0"]
end
subgraph two [Results]
GO_ana ---> GO_plot[/"GO:BP plot"/]
KEGG_ana ---> KEGG_hsa05323[/"KEGG pathway hsa05323"/]
KEGG_ana ---> KEGG_hsa04620[/"KEGG pathway hsa04620"/]
data_ana ---> Volcano[/"Volcanoplot"/]
end
click HG38 "https://www.ncbi.nlm.nih.gov/datasets/genome/GCF_000001405.40/"
click seq_map "https://github.com/Quantum-Coder826/J2P4casusTranscriptomics/blob/main/scripts/seq_mapping.R"
click matrix "https://github.com/Quantum-Coder826/J2P4casusTranscriptomics/blob/main/scripts/count_matrix.R"
click data_ana "https://github.com/Quantum-Coder826/J2P4casusTranscriptomics/blob/main/scripts/data_ana.R"
click GO_ana "https://github.com/Quantum-Coder826/J2P4casusTranscriptomics/blob/main/scripts/GO_analysis.R"
click KEGG_ana "https://github.com/Quantum-Coder826/J2P4casusTranscriptomics/blob/main/scripts/KEGG_analysis.R"
click GO_plot "https://github.com/Quantum-Coder826/J2P4casusTranscriptomics/#Fig2"
click KEGG_hsa05323 "https://github.com/Quantum-Coder826/J2P4casusTranscriptomics/#Fig3"
click KEGG_hsa04620 "https://github.com/Quantum-Coder826/J2P4casusTranscriptomics/#Fig4"
click Volcano "https://github.com/Quantum-Coder826/J2P4casusTranscriptomics/#Fig1"
click dataset "https://github.com/Quantum-Coder826/J2P4casusTranscriptomics/tree/main/dataset"
Figuur 5: Flowchart dataanalyse, alle vierkanten zijn scripts alle cylinder data en parallelogramen resultaten. Alle script bevatten core gebuikte packages en linken naar hun corresponderende bron/script.
Resultaten
De identificatie van signifikant upregulerende genen.
Uit de transcriptomische analyse zijn 2085 genen geïdentificeerd met een significante verhoging in expressie (Padj < 0.05). Zie Figuur1 voor de volcanoplot van alle geïdentificeerde genen. Tabel2 toont de 10 genen met de hoogste log2FoldChange.
 Figuur1: Volcanoplot van alle geïdentificeerde genen: gezonde personen versus RA-patiënten. Rode punten: Padj < 0.05. Grijze punten: FoldChange < 1.
Figuur1: Volcanoplot van alle geïdentificeerde genen: gezonde personen versus RA-patiënten. Rode punten: Padj < 0.05. Grijze punten: FoldChange < 1.
Tabel2: Top 10 genen (Padj < 0.05), gesorteerd op log2FoldChange. De meeste genen zijn betrokken bij de productie van immunoglobulinen (antilichamen).
| gene name | baseMean | log2FoldChange | lfcSE | stat | pvalue | padj |
|---|---|---|---|---|---|---|
| IGHV3-53 | 358.2872 | 11.42516 | 1.276373 | 8.951274 | 3.514037e-19 | 2.060776e-16 |
| IGKV1-39 | 311.7985 | 11.21278 | 1.410378 | 7.950196 | 1.862163e-15 | 4.171873e-13 |
| IGKV3D-15 | 286.7887 | 11.09350 | 1.382754 | 8.022754 | 1.034005e-15 | 2.514271e-13 |
| IGHV6-1 | 346.5721 | 10.77700 | 1.411903 | 7.632961 | 2.294216e-14 | 4.121114e-12 |
| IGHV1-69 | 180.6577 | 10.44191 | 1.253843 | 8.327928 | 8.226953e-17 | 2.523650e-14 |
| IGHV3-15 | 842.9854 | 10.42893 | 1.828176 | 5.704554 | 1.166483e-08 | 5.149668e-07 |
| IGKV1D-13 | 144.2083 | 10.11700 | 1.212725 | 8.342370 | 7.281642e-17 | 2.268573e-14 |
| IGKV2-28 | 2205.2062 | 10.04342 | 2.028891 | 4.950203 | 7.413624e-07 | 1.759765e-05 |
| IGHV4-31 | 136.0188 | 10.00943 | 1.551437 | 6.451719 | 1.105884e-10 | 8.416118e-09 |
| IGHV1-69-2 | 130.5220 | 9.91000 | 2.507762 | 3.951730 | 7.758815e-05 | 8.840172e-04 |
Identificatie van biologische processen gerelateerd aan geidentficeerde genen.
Voor de GO-analyse zijn 131 genen geselecteerd met een Padj < 0.01 en een log2FoldChange > 6.
Deze analyse identificeerde 52 biologische processen (
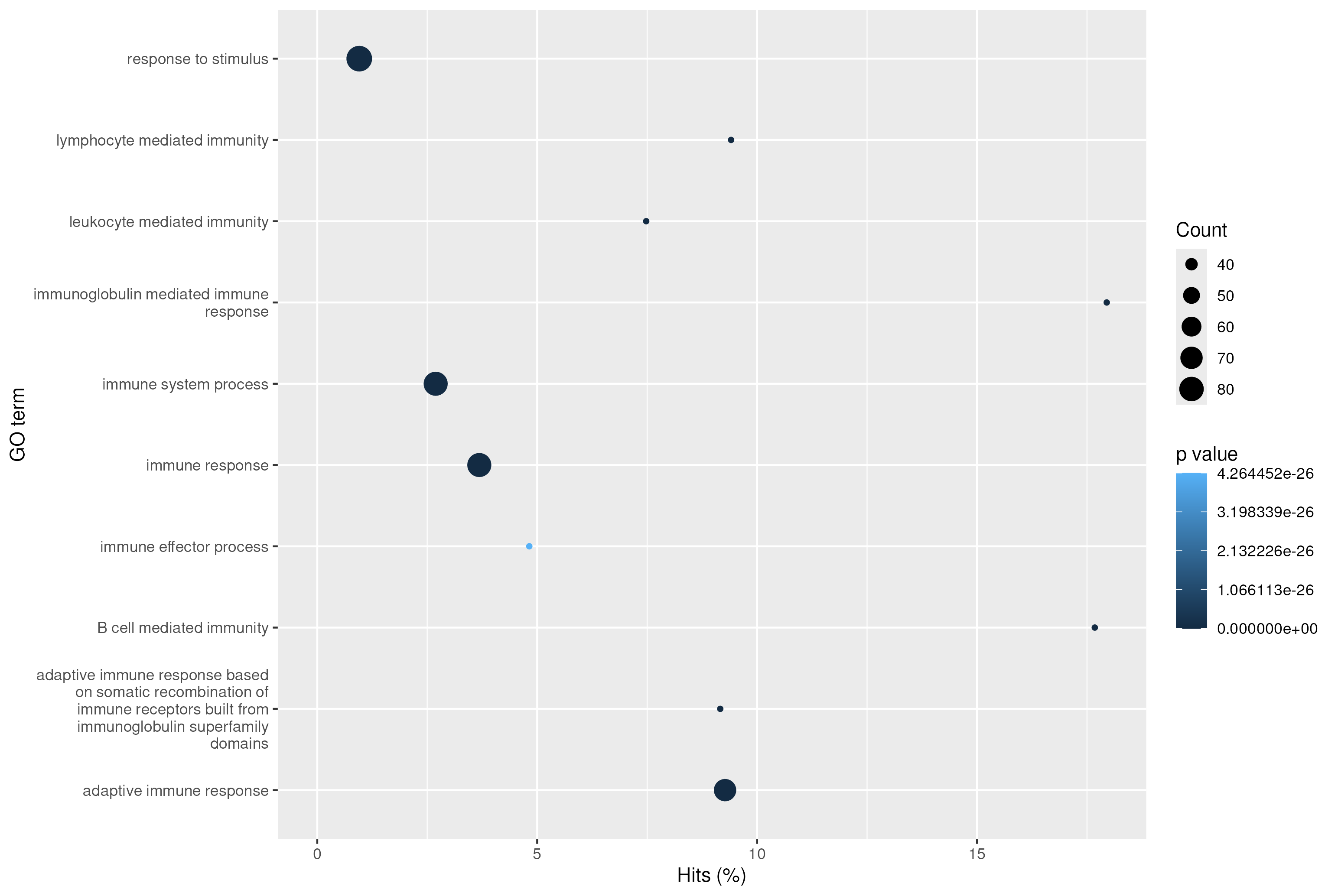 Figuur2: Gene Ontology analyse van Biological Process (BP) afgebeeld zijn eerste 10 van alle gevonden BPs
Figuur2: Gene Ontology analyse van Biological Process (BP) afgebeeld zijn eerste 10 van alle gevonden BPs
Analyse van pathways relevant tot geidentificeerde biologische processen.
Op basis van de GO-resultaten zijn de pathways hsa05323 & hsa04620 geïdentificeerd.
- Hsa05323 beschrijft in het algemeen RA-processen (Figuur3).
- Hsa04620 betreft de Toll-like receptor signaling pathway (Figuur4), betrokken bij het initiëren van inflammatoire immuunreacties.
Veel van de verhoogd tot expressie gebrachte genen hebben een inflammatoire werking, zoals TGFa, IL1, IL6 (kan ook anti-inflammatoir zijn) & IFNa[4,5].
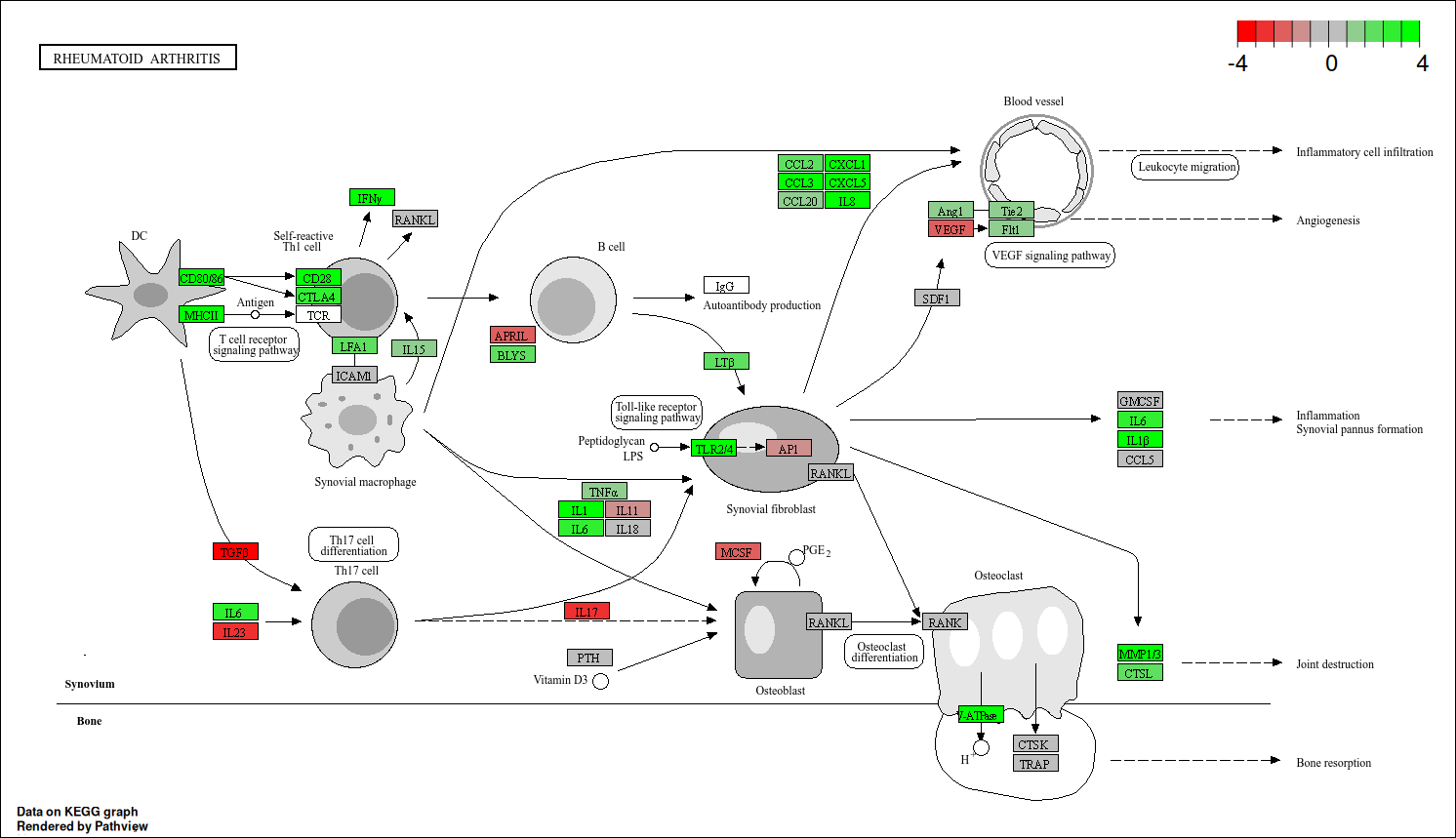 Figuur3: KEGG ziekte pathway diagram hsa05323, de algemene processen betrokken bij RA. Groen gekleurde boxen zijn up-regulerende genen rode down-regulernd. Het overgroot gedeelte van de up-gereguleerde genen stimuleert een immuunrespons.
Figuur3: KEGG ziekte pathway diagram hsa05323, de algemene processen betrokken bij RA. Groen gekleurde boxen zijn up-regulerende genen rode down-regulernd. Het overgroot gedeelte van de up-gereguleerde genen stimuleert een immuunrespons.
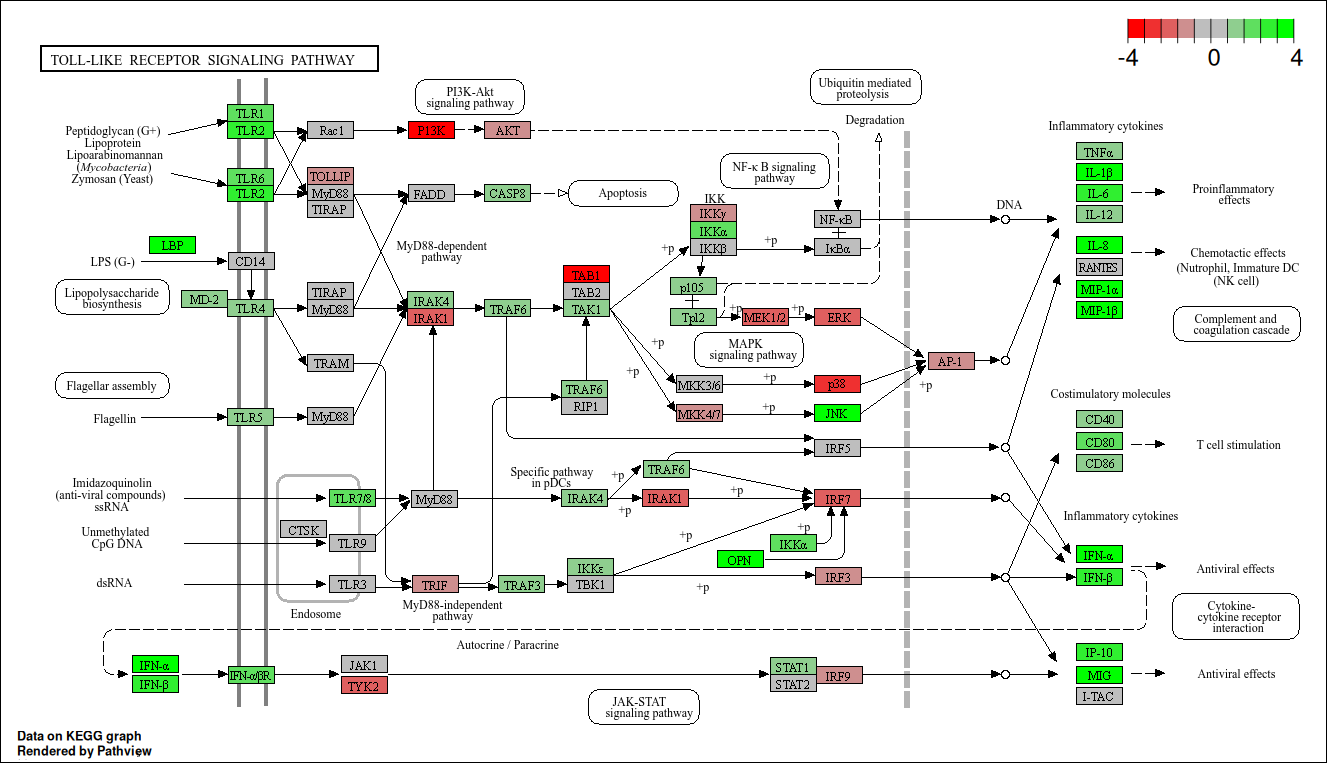 Figuur4: KEGG ziekte pathway diagram hsa04620, Toll-like signaling patyway. Deze pathway is betrokken bij het stimuleren van het immuun systeem. Het produceert voornamelijk cytokines die een inflammatry immuunrespons stimuleert. Deze genen zijn allemaal aan het up reguleren in de pathway (Groen gemarkeerd).
Figuur4: KEGG ziekte pathway diagram hsa04620, Toll-like signaling patyway. Deze pathway is betrokken bij het stimuleren van het immuun systeem. Het produceert voornamelijk cytokines die een inflammatry immuunrespons stimuleert. Deze genen zijn allemaal aan het up reguleren in de pathway (Groen gemarkeerd).
Conculsie
De vraag of er een significante verhoging is van genexpressie bij personen met Reumatoïde Artritis is beantwoord door het transcriptoom van 8 personen (4 gezond, 4 met RA) te analyseren.
Personen met RA vertonen een verhoogde inflammatoire activiteit van het immuunsysteem (Figuur 2), blijkend uit verhoogde expressie van genen die betrokken zijn bij de productie van antilichamen en pro-inflammatoire cytokinen (Figuren 3-4). Deze resultaten komen overeen met de literatuur[4,5].
Bronnen
[1] Aletaha, D., & Smolen, J. S. (2018). Diagnosis and Management of Rheumatoid Arthritis. JAMA, 320(13), 1360. https://doi.org/10.1001/jama.2018.13103
[2] Majithia, V., & Geraci, S. A. (2007). Rheumatoid Arthritis: Diagnosis and Management. The American Journal of Medicine, 120(11), 936–939. https://doi.org/10.1016/j.amjmed.2007.04.005
[3] Platzer, A., Nussbaumer, T., Karonitsch, T., Smolen, J. S., & Aletaha, D. (2019). Analysis of gene expression in rheumatoid arthritis and related conditions offers insights into sex-bias, gene biotypes and co-expression patterns. PLOS ONE, 14(7), e0219698. https://doi.org/10.1371/journal.pone.0219698
[4] Feldmann, M., Brennan, F. M., & Maini, R. N. (1996). Role of cytokines in rheumatoid arthritis. Annual Review of Immunology, 14(Volume 14, 1996), 397–440. https://doi.org/10.1146/ANNUREV.IMMUNOL.14.1.397/CITE/REFWORKS
[5] Mateen, S., Zafar, A., Moin, S., Qayyum Khan, A., & Zubair, S. (2016). Understanding the role of cytokines in the pathogenesis of rheumatoid arthritis. https://doi.org/10.1016/j.cca.2016.02.010
[6] Oliver, J. E., & Silman, A. J. (2006). Risk factors for the development of rheumatoid arthritis. Scandinavian Journal of Rheumatology, 35(3), 169–174. https://doi.org/10.1080/03009740600718080
GitHub gebuik
Voor deze casus ben ik geinstueerd om gebruik te maken van GitHub, om mijn biologische data-analyses zo repoduceerbaar mogelijk te maaken. GitHub is een platform waarop je code kan bewaren & beheren met GIT, het omschrijft zichzelf als volgd: “Git is a free and open source distributed version control system designed to handle everything from small to very large projects with speed and efficiency.” De software was ontwikkeld door Linus Torvalds voor zijn linux kernel project, nu is het de meest gebuikte versie controle systeem wereldwijd.
Ter het repoduceerbaar maken van de uitgevoerde data-analyses heb ik een lijst/flowchart gemaakt van het hele process en in de repo gezet. Deze flowchart linkt naar de scrips die ik gebuikt heb, deze scripts heb ik voorzien van comment en een logisch structuur. Bij het maaken van deze scripts heb ik met taakenlijstjes gewerkt van wat ik nodig heb en deze “Featues” stap voor stap geprogarmeerd, en deze stappen zijn door git gedocumenteerd in commits met logische messages.
Git is bedoeld om code te syncroniseren tussen meerder machines dus dat heb ik ook gedaan, on-the-go heb ik mijn laptop gebuikt en thuis of bij de grote analyse stappen mijn desktop.
Ter ondersteuning hiervan heb ik script: libs.R aangemaakt deze instaleerd alle R-packages gebuikt in dit project,
en maakt het veel makkelijker om de repo op meerdere machines te zetten, helaas heb ik niet nagedacht over versies. Ook realiseerder ik later dat bestaat Renv die dit probleem veel beter oplost.
Terwijl ik bezig was heb ik ook nagedacht over de meest logische indeling voor de repo, daar is uiteindelijk het stuctuur hieronder uitgekomen. Sommige van deze file zitten niet in de repo, dit is omdat ik die in de .gitignore heb gezet, deze file omschrijft welke files en folders niet in de repo moeten belanden.
Mappen
./bamsDeze map bevat alle Binary Alignment Maps, zowel de gesorteerde als de niet-gesorteerde varianten. Ze dienen voornamelijk als back-up../datasetBevat de .fasta-bestanden die de paired-end reads van de sequencingdata vormen. Ook dit is bedoeld als back-up../resultsHier worden alle gegenereerde resultaten opgeslagen../results/GOBevat de GOseq-resultaten, die ik apart wil bewaren../results/KEGGpathview downloadt gegevens van KEGG en dumpt deze normaal gesproken in de working directory van R. Ik wil die rommel liever ergens anders opslaan. Let op: dit is hulpdata en staat dus niet in de repo.
./scriptsBevat alle scripts die zijn gebruikt voor het genereren en analyseren van de resultaten. Het script./scripts/lib.Ris handig omdat hierin alle gebruikte R-packages zijn opgenomen en automatisch geïnstalleerd kunnen worden op de machine van de gebruiker../refSeqHomoSapiensBevat het geïndexeerde humane referentiegenoom. Let op: GitHub staat geen bestanden toe groter dan 100 MB. Deze map bevat ±23 GB aan data en is daarom niet geüpload.